En los últimos años se ha generado una ingente cantidad de datos genómicos, transcriptómicos y proteómicos en organismos de relevancia para las ciencias del vino, como vid, levaduras, microorganismos de vinificación, organismos de suelo o el propio ser humano. El análisis e interpretación de dichos datos a nivel molecular es fundamental para su aplicación racional a la selección y mejora de organismos o al diseño de nuevas entidades moleculares de interés biotecnológico y fitosanitario. Para ello, es necesario conocer la estructura a nivel atómico de biomoléculas esenciales como proteínas y ácidos nucleicos, así como la de sus interacciones, que controlan procesos esenciales como la síntesis de proteínas, señalización celular, o replicación y reparación de ADN. El problema es que en estos momentos no existen datos estructurales para la inmensa mayoría de los complejos proteína-ADN existentes, ya que su obtención experimental es un proceso costoso y muchas veces incierto, lo que supone una gran limitación en cuanto a la interpretación de los datos genómicos. Por ello, es necesario disponer de herramientas computacionales fiables que puedan estimar la estructura de dichos complejos y así complementar los escasos datos experimentales.
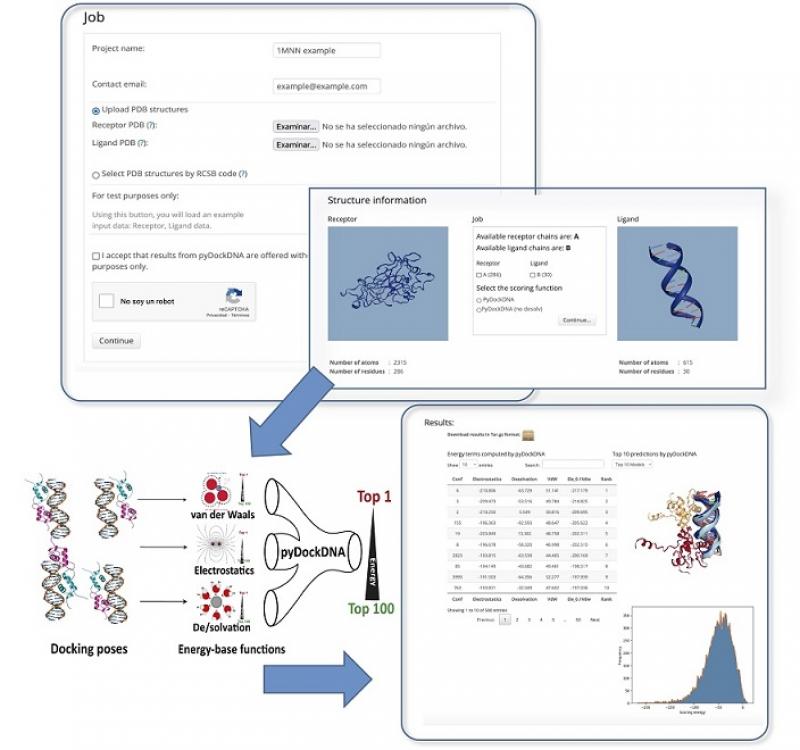
En el ICVV, se ha desarrollado una nueva herramienta en bioinformática estructural llamada pyDockDNA que permite modelar complejos entre proteína y ADN mediante técnicas avanzadas de búsqueda de orientaciones inter-moleculares de altas prestaciones y descripción eficaz de su energía de unión. Esta herramienta se ha implementado como un servidor web (https://model3dbio.csic.es/pydockdna) abierto a la comunidad científica internacional, para poder realizar estos cálculos de modelado molecular de forma automática, mediante un uso racional de los recursos computacionales del centro. Este servidor web contribuirá al portafolio de herramientas y servicios previstos para la futura plataforma de Bioinformática, Modelización y Análisis de Datos Biológicos que se prevé poner en marcha en el centro.
Este trabajo ha sido llevado a cabo por los investigadores Juan Fernández Recio y Luis Angel Rodríguez Lumbreras del grupo de Bioinformática Estructural (3DBIOwine) del Instituto de Ciencias de la Vid y del Vino, junto con otros investigadores de la Universidad de Valencia y de la empresa Zymvol Biomodeling SL.
Los resultados se han publicado en el siguiente artículo:
Rodríguez-Lumbreras LA, Jiménez-García B, Giménez-Santamarina S and Fernández-Recio J (2022) pyDockDNA: A new web server for energy-based protein-DNA docking and scoring. Front. Mol. Biosci. 9:988996. DOI: 10.3389/fmolb.2022.988996
https://www.frontiersin.org/articles/10.3389/fmolb.2022.988996